Inhalt
Reverse Clinical Engineering®
Mit In-vitro-Diagnostik an Patienten-abgeleiteten 3D-Tumormodellen zur modernen Präzisionsonkologie
Mit dem Reverse Clinical Engineering® (RCE) hat ASC Oncology ein ergänzendes Testverfahren entwickelt, mit dem überprüft werden kann, welche medikamentösen Krebstherapien beim individuellen Patienten mit malignem soliden Tumor mit hoher Wahrscheinlichkeit wirken und welche nicht. Die Methode zählt zu den In-vitro-Diagnostika und basiert auf Patienten-abgeleiteten 3D (PD3D®)-Zellkulturen, sogenannten Tumororganoiden, die aus einer vitalen Tumorprobe des Patienten gewonnen werden. Durch das RCE-Testverfahren stehen dem behandelnden Arzt zusätzliche Daten zur Verfügung, die es ihm und dem Patienten erleichtern eine optimale Therapieentscheidung zu treffen. So kann so früh wie möglich eine personalisierte Therapie mit voraussichtlich höheren Überlebenschancen und besserer Lebensqualität ausgewählt werden sowie eine wahrscheinlich unwirksame Behandlung samt potentieller ungewollter Nebenwirkungen vermieden werden.
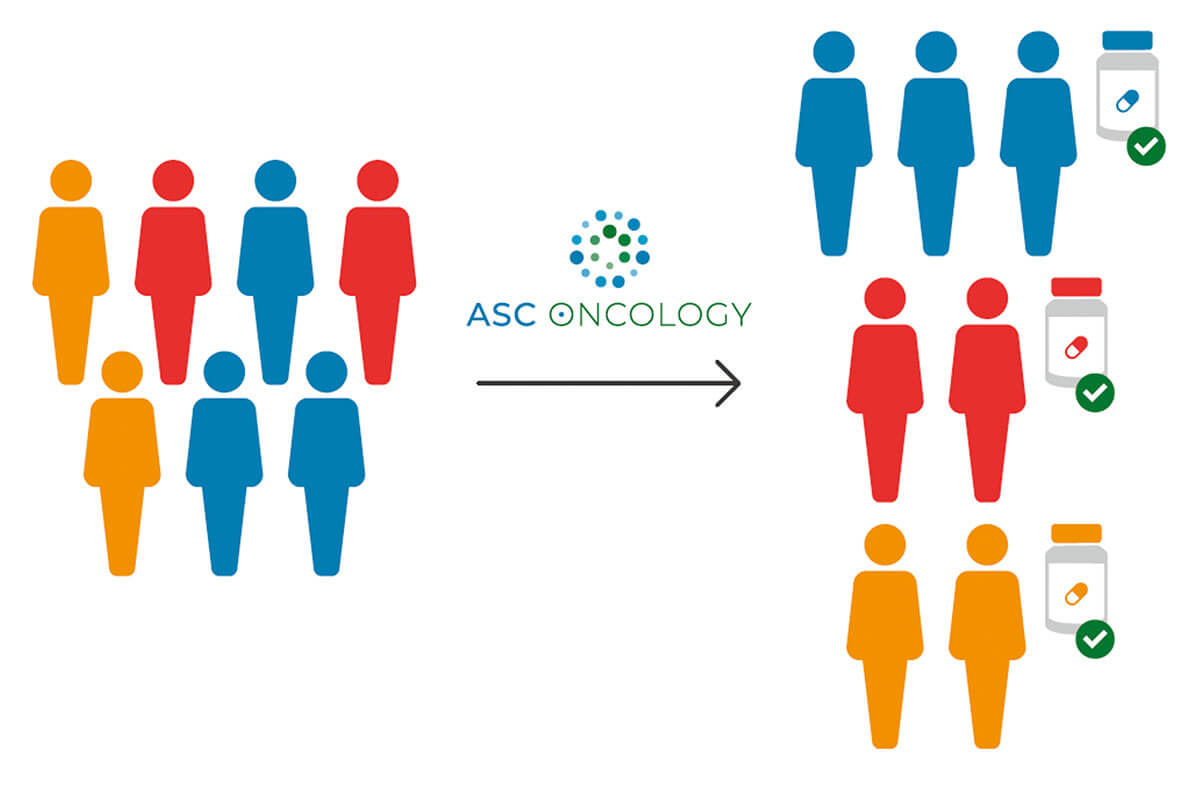
Medizinischer Bedarf
Tumore weisen eine erhebliche Heterogenität auf, was eine Herausforderung für die Behandlung darstellt (Burrell et al., 2013): während viele Tumore auf empfohlene Standardtherapieregime ansprechen, gibt es einen bedeutenden Anteil an Non-Respondern, bei denen etablierte Therapiekonzepte nicht die erhoffte Wirkung zeigen. Indem das RCE-Testverfahren ergänzende Daten für die Therapieentscheidung zur Verfügung stellt, werden Ärzte dabei unterstützt, ihren Patienten die am besten geeignete Therapie mit den höchsten Erfolgschancen anbieten zu können.
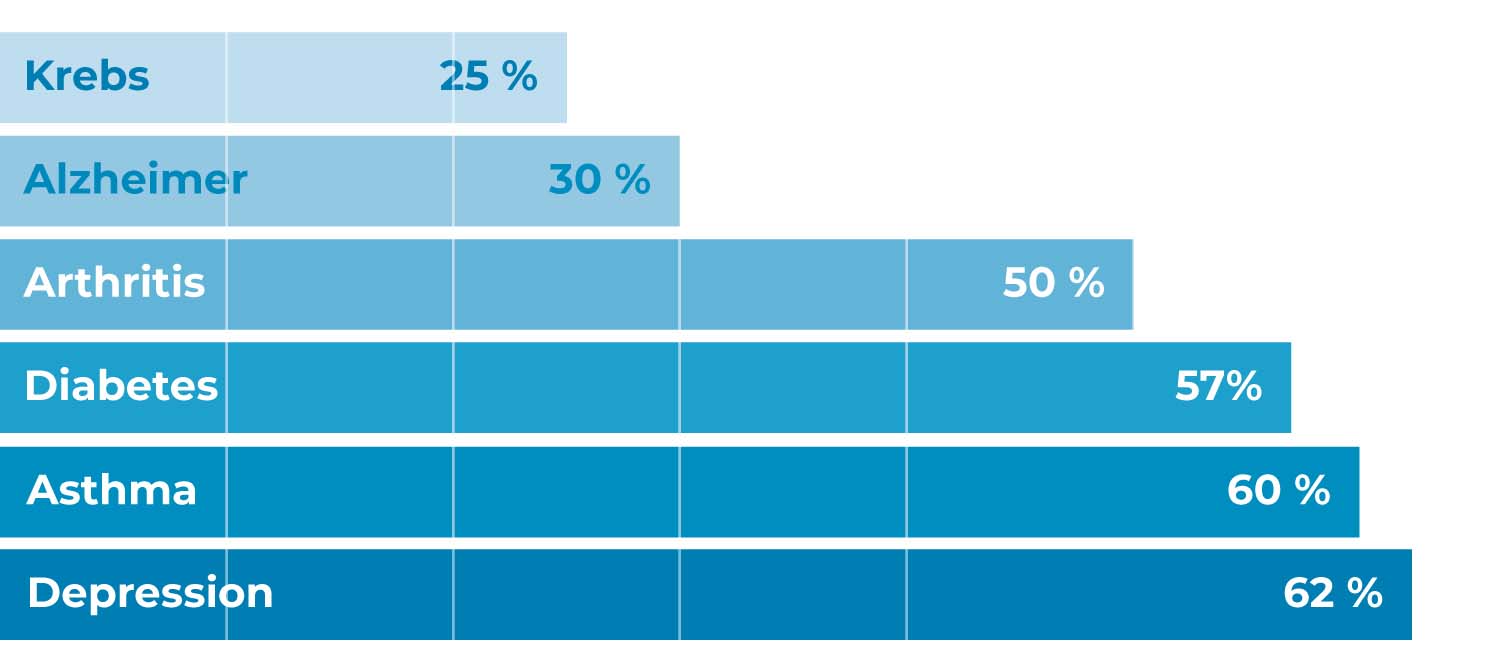
Anteil von Patienten, bei denen ein verabreichtes Medikament effektiv ist, für ausgewählte Erkrankungen (Statista, 2021).
Reverse Clinical Engineering® + klinische Studiendaten = mehr Erkenntnisse
Das Testverfahren stellt eine wichtige Ergänzung zu randomisierten kontrollierten Studien (RCTs) dar. Im Sinne einer n = 1-Studie ermöglicht es die Wirksamkeit von medikamentösen Therapien für den einzelnen Patienten ganz individuell vor Therapiebeginn zu bestimmen. Hingegen können RCTs die generelle Wirksamkeit und Sicherheit einer Therapie untersuchen. Die Aussagekraft für den einzelnen Patienten bleibt dabei jedoch begrenzt, da sich aus den ermittelten Durchschnittswerten großer Kohortenstudien nur bedingt das individuelle Ansprechen ableiten lässt.

King’s College London & University College London Cancer Institute
Mit dem Reverse Clinical Engineering® auf dem Weg zu einer verbesserten individuellen Therapieentscheidung
Personalisierte Therapiekonzepte und Stratifizierungsmöglichkeiten nach molekularen Tumorsubtypen finden zunehmend Einzug in den klinischen Alltag. Jedoch steht die Präzisionsmedizin noch am Anfang: Studien ergaben, dass mithilfe von Sequenzierungen bei weniger als der Hälfte der Patienten therapeutisch verwertbare Treibermutationen ausfindig gemacht werden konnten und lediglich 0,8-3 % der Patienten anschließend von einer abgestimmten Therapie profitieren konnten (Tannock & Hickman, 2019).
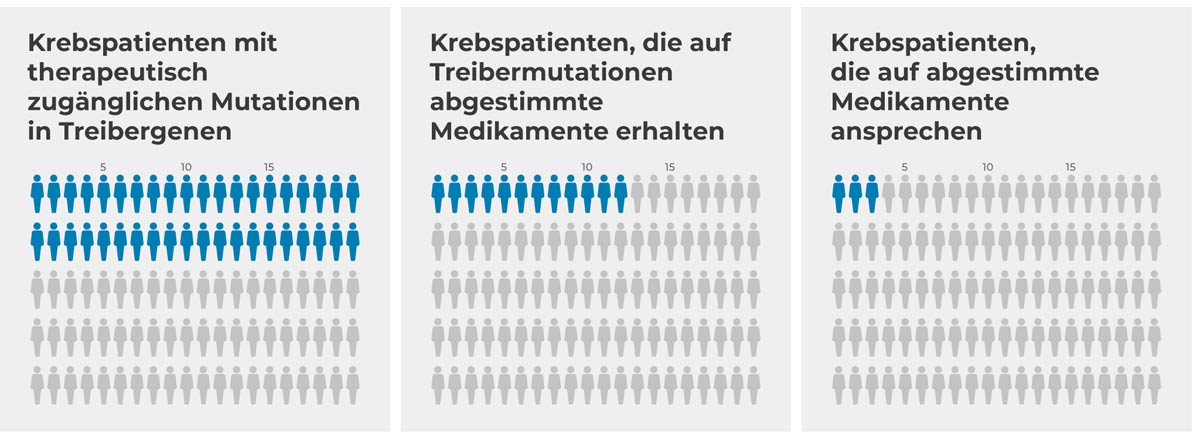
Nur ein kleiner Anteil der Patienten, die sich einem Gentest unterzogen haben und daraufhin mit einer auf eine Mutation abzielenden Therapie behandelt wurden, konnten von der zielgerichteten Behandlung profitieren (Tannock & Hickman, 2019).
Die wissenschaftliche Evidenz dafür wächst, dass Tumore eine Heterogenität bzw. Plastizität aufweisen, die über die genetische Ebene hinausreicht (Brock & Huang, 2017). Der Zellverbund und die Mikro-Umgebung scheinen Einfluss darauf zu haben, wie der Tumor auf eine Behandlung reagiert und ob beispielsweise Resistenzen entstehen (Huang, 2021). Wie die Plastizität das Therapieansprechen des Tumors beeinflusst, lässt sich durch Genom-Sequenzierung oder andere Formen der Biomarkerbestimmung bisher noch ungenügend abbilden. Ein Teil davon kann in Patientenabgeleiteten 3D-Tumormodellen jedoch erhalten werden (Walter et al., 2021, Yao 2020, Yang et al., 2022) – weshalb das Testverfahren eine wertvolle Ergänzung in der Diagnostik darstellt.

Reverse Clinical Engineering® um das Risiko weiterer Therapielinien zu reduzieren
Mit jeder weiteren Therapielinie verschlechtert sich die Prognose für den Patienten, da die Wahrscheinlichkeit eines Therapieansprechens deutlich abnehmen kann (Kessous et al., 2020). Das RCE-Testverfahren hilft dabei frühzeitig die bestmögliche Therapieoption zu identifizieren, sodass das Risiko einer unwirksamen Behandlung sowie daraus resultierender weiterer Therapielinien minimiert werden kann. Auch bei seltenen oder austherapierten Tumoren, für die Leitlinienempfehlungen oft nur mit geringem Evidenzgrad vorliegen, kann das Testverfahren helfen rechtzeitig die richtige Therapieoption zu identifizieren.
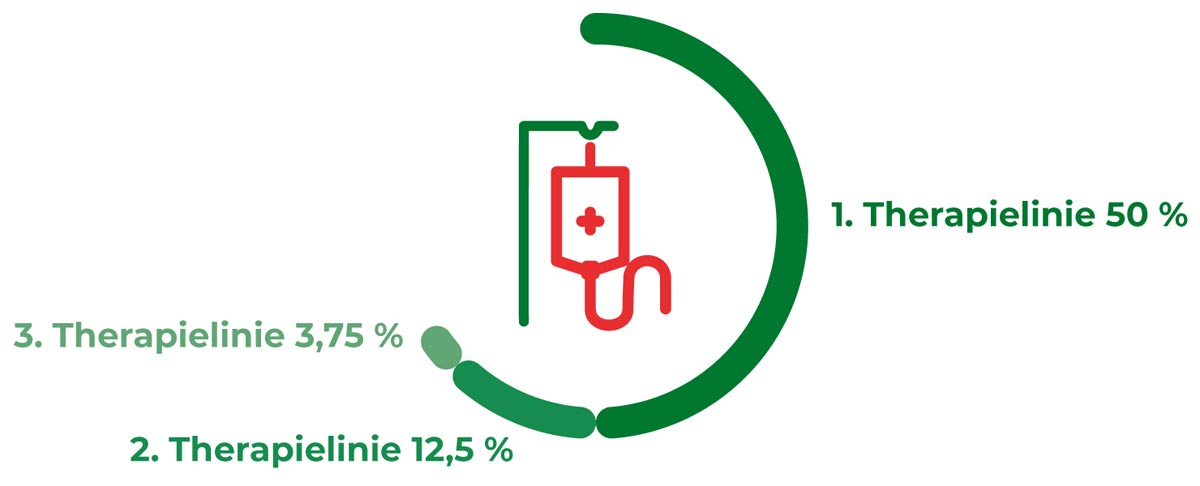
Die Ansprechraten von soliden Tumoren nehmen mit fortschreitender Therapielinie ab (Regenbrecht et al., 2018).
Funktionsweise des Testverfahrens
Das RCE-Testverfahren basiert auf PD3D®-Tumormodellen, sogenannten Organoiden. Unsere Wissenschaftler haben gemeinsam mit Partnern aus der Pharmaforschung eine automatisierte Plattform entwickelt, mit der Tumororganoide im Hochdurchsatz parallelisiert und standardisiert untersucht werden können (Boehnke et al., 2016). Das Verfahren hat beim National Cancer Institute (NCI) den Einsatz des NCI60 Panels zur prä-klinischen Medikamententestung inzwischen vollständig abgelöst und wurde vom NCI als „best practice approach“ gekürt (Ledford, 2016, Boehnke et al., 2016, NCI, 2019).

King’s College London & University College London Cancer Institute
Von der frischen Gewebeprobe zum Patienten-abgeleiteten 3D-Tumormodell
PD3D®-Tumormodelle können aus frischen Gewebeproben wie Resektaten oder Biopsien gewonnen werden. Dafür wird die Probe im Labor mechanochemisch aufbereitet und lebende Tumorzellen anschließend in einer Matrix kultiviert. Diese ähnelt der menschlichen extrazellulären Basalmembran und imitiert so die komplexe Umgebung des Tumors. Die Matrix bildet außerdem ein Hydrogel, dass es den Zellen erlaubt dreidimensional zu wachsen. Zudem wird jede einzelne Tumorentität in einem eigens optimierten Medium kultiviert, dass das Wachstum der Tumorzellen begünstigt und die Vermehrung anderer nicht entarteter Zellen der Probe begrenzt. So können sich aus den Tumorzellen Organoide entwickeln, die aus organspezifischen Zelltypen aufgebaut sind und räumlich ähnlich organisiert sind, wie im Ursprungsorgan des Tumors.
Die Organoide weisen genomische, molekulare und phänotypische Eigenschaften des Patiententumors auf, die auch über mehrere Passagen in Langzeitkulturen stabil aufrechterhalten werden können (Sachs et al.,2018). Als präzise und klinisch relevante Kopien, können sie auch verschiedene Subtypen und die intratumorale Heterogenität des Patiententumors abbilden (Kopper et al., 2019, Sharick et al., 2020).
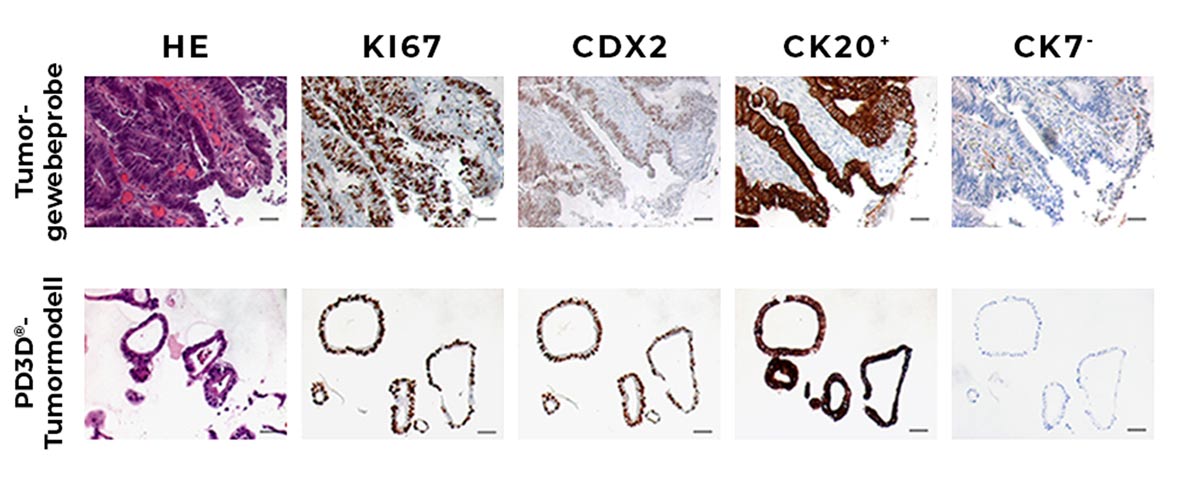
Vergleich zwischen der immunhistologischen Färbung eines Primärtumors und dem abgeleiteten PD3D®-Tumormodell, gefärbt für Hämatoxylin-Eosin (HE), CDX2, CK20+ und CK7–. Die Ki67-Expression markiert aktiv proliferierende Zellen sowohl im Gewebe als auch im Modell (Schütte et al., 2017).
Von der Substanztestung bis zur Erstellung des Sensitivitätsprofils
Nachdem die PD3D®-Modelle angelegt und vervielfältigt wurden, können die infrage kommenden medikamentösen Therapien getestet werden. Dafür werden die Tumororganoide in 96- oder 384-Well-Platten kultiviert, automatisiert und parallelisiert mit den zu testenden Substanzen behandelt und anschließend die Zellviabilität gemessen und ausgewertet.
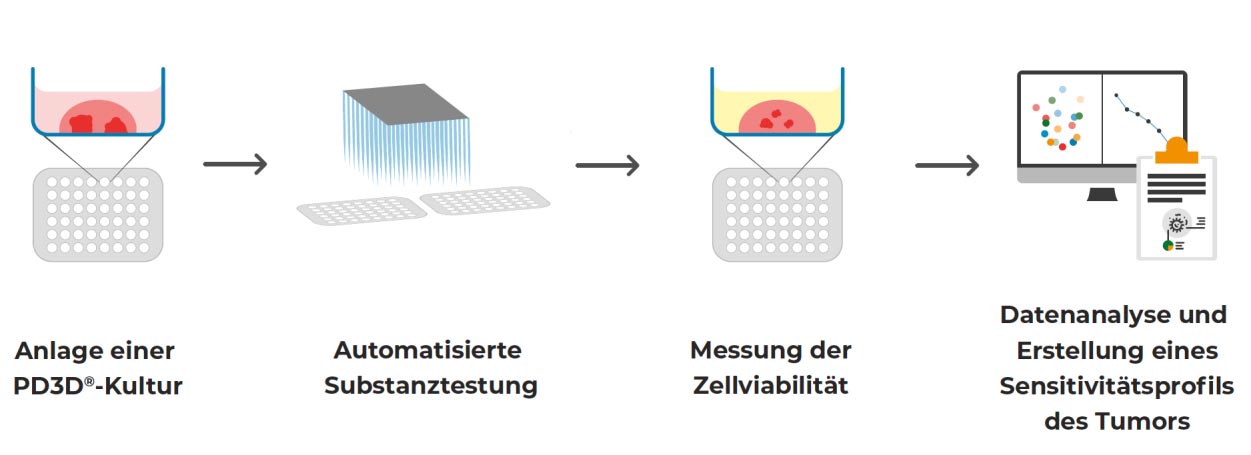
Durchführung des Reverse Clinical Engineerings®
Um klinisch relevante Ergebnisse zu erzielen, werden die Wirkstoffe in unterschiedlichen Konzentrationen untersucht, die sich an der klinisch messbaren maximalen Konzentration (Cmax) des jeweiligen Wirkstoffs im Blutplasma orientieren. Die Zellviabilität wird mittels eines ATP-abhängigen Lumineszenz-Assays bestimmt. Dabei wird der ATP-Gehalt der behandelten Tumororganoide quantifiziert, welcher direkt proportional zur Anzahl der stoffwechselaktiven, lebensfähigen Zellen ist (Crouch et al., 1993).
Zeitrafferaufnahmen von PD3D®-Tumormodellen ohne Substanztestung (Control) und mit Staurosporin-Behandlung, welche in steigender Konzentration (50 pM < 5 nM < 0,5 µM) zum vermehrten Zelltod führt.
Anhand der Daten lässt sich anschließend der IC50-Wert ermitteln, der angibt, welche Wirkstoffkonzentration zur halbmaximalen Wachstumshemmung der Tumorzellen führt. Liegt der IC50-Wert oberhalb des Cmax-Wertes (IC50 > Cmax) ist es unwahrscheinlich, dass die Therapie beim Patienten zu einem effektiven Tumoransprechen führt. Ein niedrigerer IC50, der unterhalb des Cmax liegt (IC50 < Cmax), wird mit größerer Wahrscheinlichkeit das Wachstum der Tumorzellen hemmen und zu einem Therapieansprechen führen.
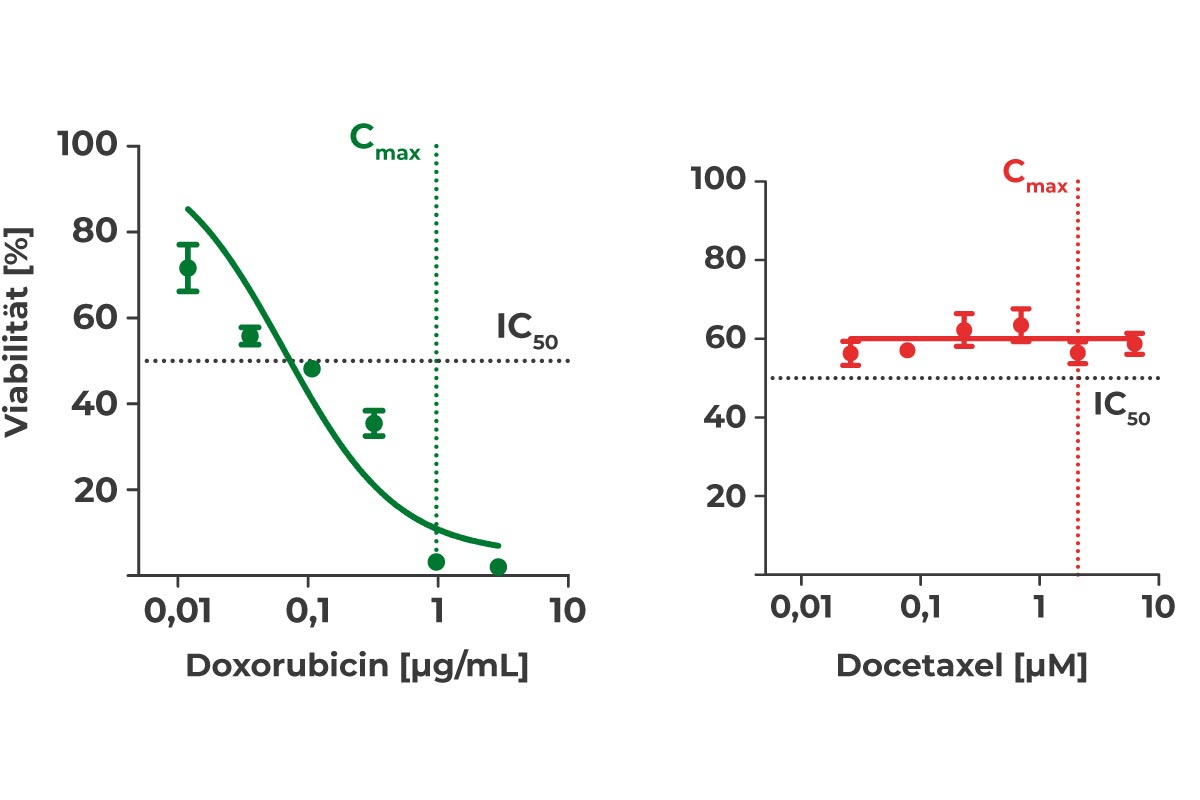
Dosis-Wirkungskurven für Doxorubicin (grün: IC50 < Cmax) und Docetaxel (rot: IC50 > Cmax).
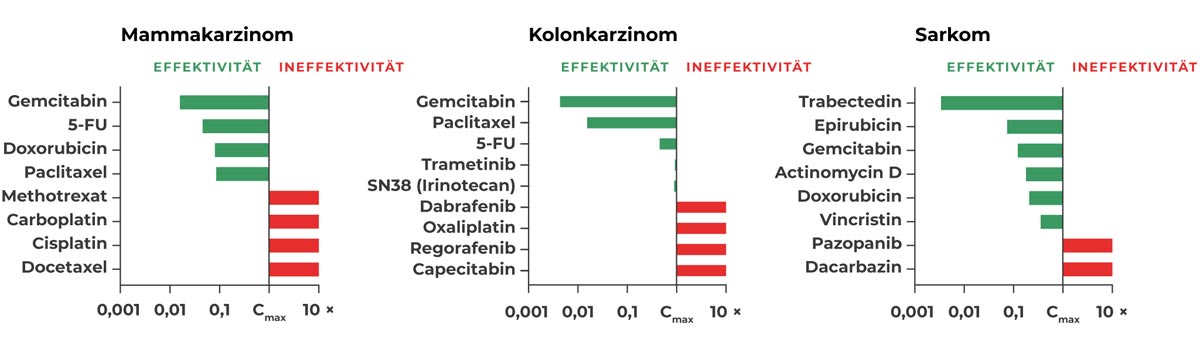
Ansprechen verschiedener PD3D®-Tumormodelle auf ausgewählte Substanzen orientiert am klinisch maximalen Plasmaspiegel (grün: IC50 < Cmax, rot: IC50 > Cmax).
Abschließend erstellen wir in durchschnittlich 28 Tagen nach Probeneingang einen Bericht, der eine detaillierte Auswertung und grafische Aufbereitung der Daten sowie konkrete Empfehlungen für die nachfolgende Therapieentscheidung enthält. Wichtig: Die Empfehlung sollte immer im Zusammenhang mit weiteren Daten im klinischen Gesamtbild interpretiert werden und als wertvolle, ergänzende Entscheidungshilfe für die Therapieplanung mit dem Patienten verstanden werden.
Performance
Die Anwendung von Tumororganoiden für eine verbesserte personalisierte Therapie wurde in internationalen ko-klinischen Studien demonstriert. Das Verfahren wird zudem vom NCI seit 2017 als „best practice approach“ zur Medikamententestung an Tumororganoiden empfohlen und hat sich in der Präklinik bereits etabliert (Boehnke et al., 2016, NCI, 2019). Zudem bereitet ASC Oncology eine multizentrische internationale Phase IV Studie in der Erstlinientherapie von schwer behandelbaren Tumorerkrankungen mit der Medizinischen Universität Innsbruck als leitendes Prüfzentrum vor.
Medizinische Statistik
Eine Metaanalyse 17 ko-klinischer Studien (u.a. beim kolorektalen Karzinom, Rektalkarzinom, Magenkarzinom, Pankreaskarzinom, Ovarialkarzinom und Mammakarzinom; Wensink et al., 2021) ergab folgende Spezifikationen für das Testverfahren an Tumororganoiden:
Diagnostische Sensitivität: 81 % (95 %-KI: 0,69 – 0,89)
Diagnostische Spezifität: 74 % (95 %-KI: 0,64 – 0,82)
Bei individuellen Tumorentitäten ergaben ko-klinische Studien folgende Werte:
metastasierende Krebserkrankungen des Magen-Darm-Trakts (n = 15)
Diagnostische Sensitivität: 100 %
Diagnostische Spezifität: 93 %
Positiver prädiktiver Wert: 88 %
Negativer prädiktiver Wert: 100 %
Plattenepithelkarzinome im Kopf-Hals-Bereich (n = 7)
Diagnostische Sensitivität: 75 %
Diagnostische Spezifität: 100 %
Positiver prädiktiver Wert: 100 %
Negativer prädiktiver Wert: 75 %
fortgeschrittenes kolorektales Karzinom (n = 17)
Diagnostische Sensitivität: 100 %
Diagnostische Spezifität: 86 %
Positiver prädiktiver Wert: 92 %
Negativer prädiktiver Wert: 100 %
Pankreaskarzinom (n = 9)
Diagnostische Sensitivität: 100 %
Diagnostische Spezifität: 67 %
Positiver prädiktiver Wert: 86 %
Negativer prädiktiver Wert: 100 %
fortgeschrittenes Lungenkarzinom (n = 54)
Diagnostische Sensitivität: 84 %
Diagnostische Spezifität: 83 %
Positiver prädiktiver Wert: 81 %
Negativer prädiktiver Wert: 86 %
Effektivität – höhere Chance auf ein verbessertes Therapieansprechen
Signifikant verlängertes progressionsfreies Überleben (PFS) nach Tests an patienteneigenen Tumorzellen
Bei Patienten mit fortgeschrittenem Darmkrebs konnte die Effektivität einer adjuvanten Chemotherapie nach einer Tumorresektion anhand von Organoid-basierten Tests vorausgesagt werden. Patienten, deren Tumor-Organoide im Test sensitiv für die Chemotherapie waren, hatten demnach ein deutlich längeres PFS von 16,0 Monaten als Patienten, deren Tumor-Organoide im Test resistent gegenüber der Chemotherapie waren und die ein kürzeres PFS von 9,0 Monaten aufwiesen (p < 0,001; Wang et al., 2023).
Organoid-basierte Tests wurden in einer weiteren Studie bei Patienten mit metastasiertem Darmkrebs genutzt, um das Ansprechen auf eine FOLFOX-Therapie zu überprüfen. Patienten, deren Tumor-Organoide im Test resistent gegenüber FOLFOX waren, hatten ein mehr als 4-mal so hohes Progressions- oder Sterberisiko wie Patienten, deren Tumor-Organoide im Test für die Therapie sensitiv waren (HR: 4,3; 95 %-Konfidenzinterval: 2,6-7,2; p < 0,001; Tang et al., 2023). Das mediane PFS betrug in der Organoid-resistenten Patientengruppe 8 Monate und in der Organoid-sensitiven Gruppe 11 Monate.
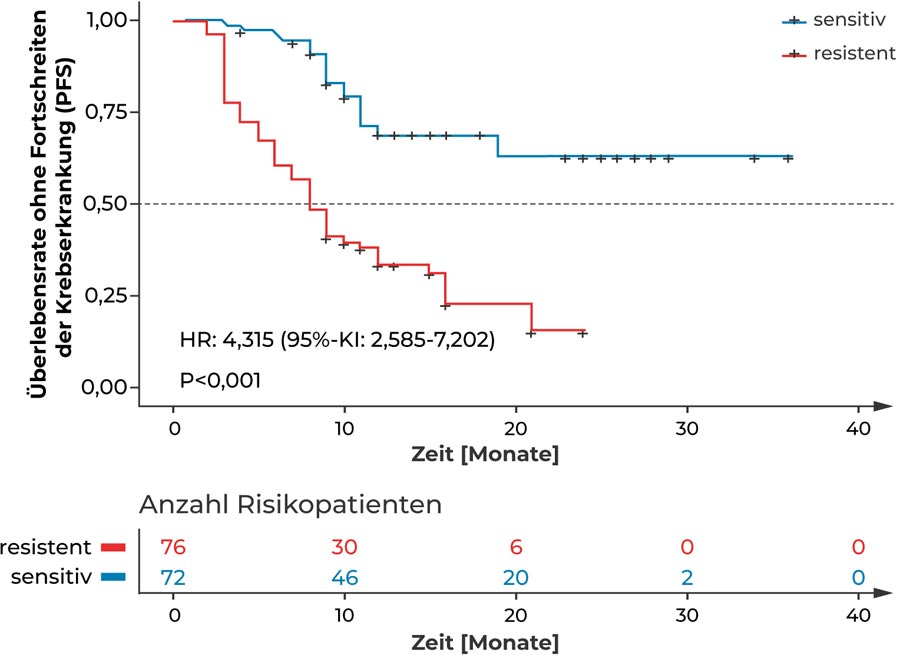
Die Kaplan-Meier-Kurven zeigen, dass Patienten mit metastasiertem Darmkrebs, die eine FOLFOX-Therapie erhielten ein signifikant höheres Progressions- oder Sterberisiko hatten, wenn ihre Organoide im Test zuvor resistent gegenüber FOLFOX waren im Vergleich zu Patienten, deren Organoide im Test sensitiv für FOLFOX waren (Tang et al., 2023). FOLFOX = Folinsäure, 5-Fluorouracil, Oxaliplatin, HR = Hazard Ratio, KI = Konfidenzintervall
Auch bei Patienten mit Lungenkrebs verlängerte sich das PFS signifikant durch den Einsatz von Tests an patienteneigenen Tumorzellen um mehr als das doppelte von 8,5 auf 18 Monate (p < 0,05; Chen et al., 2018).
Patienten mit einem Pankreaskarzinom, die basierend auf In-vitro-Tests an individuellen Tumororganoiden als „Responder“ für eine adjuvante Therapie eingestuft wurden, wiesen ein RFS > 12 Monate nach einer operativen Resektion auf, wohingegen die Erkrankung bei Patienten, die aufgrund der In-vitro-Daten als „Non-Responder“ eingestuft wurden, innerhalb eines Jahres unter der adjuvanten Therapie rezidivierte (Sharick et al., 2020).
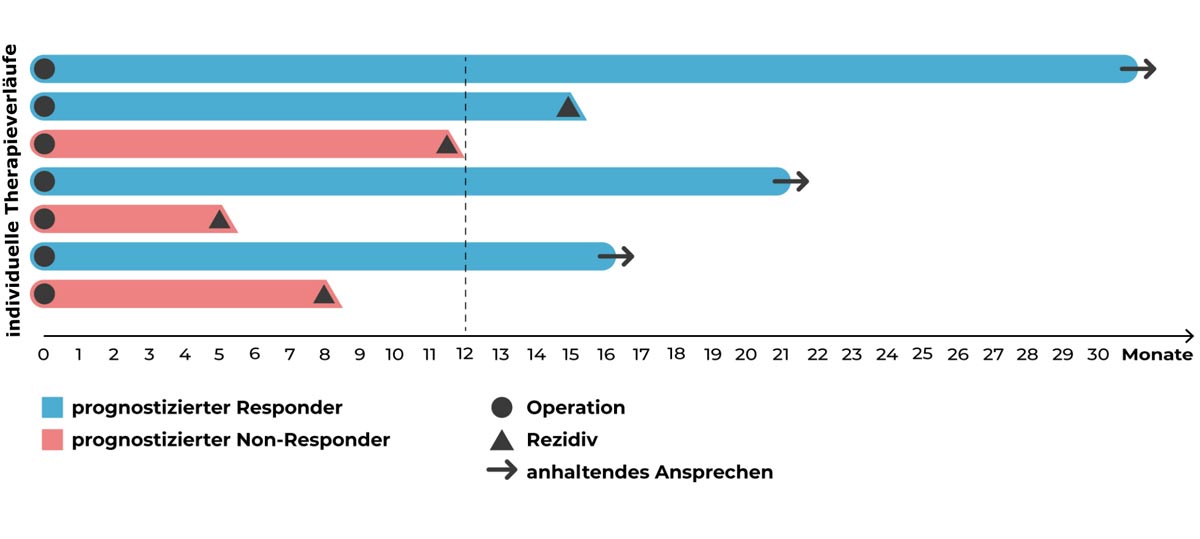
Patienten mit Pankreaskarzinom hatten ein verlängertes rezidivfreies Überleben nachdem sie basierend auf In-vitro-Tests an individuellen Tumororganoiden eine Therapie erhielten, die der Test als effektiv eingestuft hatte, verglichen mit Patienten, die eine Therapie erhielten, die der Test als ineffektiv eingestuft hatte (Sharick et al., 2020). Die Pfeile zeigen an, dass der Patient zum Zeitpunkt der Veröffentlichung der Daten weiterhin ohne Rezidiv lebte.
Lebensqualität – Verbesserte Patientenzufriedenheit
Gesteigerte Adhärenz und Vermeidung potentiell ungewollter Nebenwirkungen von wenig erfolgsversprechenden Therapien durch Präzisionsmedizin
Maßgeschneiderte Therapiepläne mit möglichst geringen Nebenwirkungen und die Teilhabe des Patienten an der Therapieentscheidung können die Adhärenz, das Vertrauen, die Versorgungsqualität und somit den Erfolg einer Krebsbehandlung begünstigen (Shingler et al., 2014, Kehl et al., 2015).
Mehrwert für das Gesundheitssystem
Indem voraussichtlich unwirksame Therapien vermieden werden, können die begrenzten Ressourcen des Gesundheitssystems geschont und kostenintensive Behandlungen effektiver eingesetzt werden. Das kann zu einer relevanten Kostenersparnis führen und personelle Kapazitäten freisetzen.
Anforderungen und Einschränkungen
Für das Testverfahren geeignetes Probenmaterial
- frisches Gewebe von malignen soliden Tumoren jeglichen Stadiums (Primärtumor oder Metastasen)
- gewonnen durch Resektion oder Biopsie
- vor oder während der laufenden Behandlung (zwischen letztem Therapiezyklus und Probenentnahme sollten mindestens zwei Wochen liegen)
Für das Testverfahren ungeeignetes Probenmaterial
- Proben von Leukämien oder Lymphomen
- konserviertes oder fixiertes Gewebe
- Gewebeproben mit zu geringer Anzahl vitaler Zellen (KEINE fixierten oder eingefrorenen Proben)
Substanzen, die für das Testverfahren geeignet sind
- Zytostatika
- zielgerichtete Therapien
- Antikörper
- Präparate im Off-Label-Use
- aktuelle klinische Prüfpräparate
- experimentelle, alternativmedizinische oder naturheilkundliche Medikamente
- je als Einzel- oder Kombinationstherapie
Substanzen, die für das Testverfahren ungeeignet sind
- Wirkstoffe, die verstoffwechselt werden müssen und deren aktiver Metabolit nicht chemisch stabil verfügbar ist
- Substanzen, die ihre Wirkung auf den Tumor ausschließlich über andere Zelltypen entfalten und eine indirekte Wirkung auf den Tumor haben
FAQ
Was unterscheidet das Reverse Clinical Engineering®-Testverfahren von anderen gängigen Tests?
Anhand der PD3D®-Tumormodelle kann noch vor Behandlungsbeginn das individuelle Therapieansprechen eines Krebspatienten in einer n=1-Studie in vitro ermittelt werden. Anhand der Daten kann in Ergänzung zu anderen klinischen Parametern eine optimale Entscheidung für eine voraussichtlich effektive Therapieoption getroffen werden.
Auch Patienten-abgeleitete Xenograftmodelle in Mäusen erlauben es, eine Voraussage über das individuelle Tumoransprechen eines Patienten zu machen. Die Modelle sind jedoch verhältnismäßig zeitaufwendig: Sie brauchen mit etwa 4-6 Monaten im Vergleich zu durchschnittlich 28 Tagen mit dem Reverse Clinical Engineering®-Testverfahren deutlich länger, um Ergebnisse zu liefern.
Sequenzierungs-Technologien können zudem den Mutationsstatus eines Tumors aufdecken, der Hinweise auf eine mögliche zielgerichtete Therapie geben kann. Ob der Tumor auf eine solche Therapie tatsächlich ansprechen wird, ist vor Behandlungsbeginn jedoch nur eingeschränkt vorhersehbar, da ohne individuelle Daten von den Durchschnittswerten großer Kohorten-Studien extrapoliert werden muss.
Wie lange dauert das Reverse Clinical Engineering®-Testverfahren?
Durchschnittlich dauert das Reverse Clinical Engineering®-Testverfahren vom Probeneingang bis zum Abschlussbericht 28 Tage. Die Dauer des Verfahrens kann je nach Tumorentität, Probenqualität und Probenmenge variieren, da diese Faktoren die Zeit beeinflussen, die zur Vermehrung der PD3D®-Tumormodelle benötigt wird.
Welche Ergebnisse liefert der Bericht des Reverse Clinical Engineering®-Testverfahrens von ASC Oncology?
Der Bericht liefert eine tabellarische Übersicht der experimentell ermittelten IC50-Werte aller getesteten Substanzen sowie eine konkrete Empfehlung für die nachfolgende Therapieentscheidung. Die Daten werden zusätzlich grafisch aufbereitet, um das individuelle Sensitivitätsprofil besser zu veranschaulichen. Zudem beinhaltet der Bericht eine ausführliche Erläuterung der Methode sowie Definitionen der wichtigsten Begriffe. Sehen Sie sich einen Beispielbericht an.
Weiterführende Literatur
Boehnke K, Iversen PW, Schumacher D, Lallena MJ, Haro R, Amat J, Haybaeck J, Liebs S, Lange M, Schäfer R, Regenbrecht CRA, Reinhard C, Valesco A: Assay Establishment and Validation of a High-Throughput Screening Platform for Three-Dimensional Patient-Derived Colon Cancer Organoid Cultures. J Biomal Screen 21, 931-41 (2016).
Diese Arbeit beschreibt die Etablierung unserer Plattform zur automatisierten und parallelisierten Medikamententestung an Organoiden.
Gaebler M, Silvestri A, Haybaeck J, Reichardt P, Lowery CD, Stancato LF, Zybarth G, Regenbrecht CRA: Three-Dimensional Patient-Derived In Vitro Sarcoma Models: Promising Tools for Improving Clinical Tumor Management. Front Oncol 11, 7 (2017)
Diese Übersichtsarbeit liefert einen umfassenden Überblick dazu wie Organoid-Modelle genutzt werden können, um die Molekularbiologie von Sarkomen besser zu verstehen.
Schütte M, Risch T, Abdavi-Azar N, Boehnke K, Schumacher D, Keil M, Yildiriman R, Jandrasits C, Borodina T, Amstislavskiy V, Worth CL, Schweiger C, Liebs S, Lange M, Warnatz HJ, Butcher LM, Barrett JE, Sultan M, Wierling C, Golob-Schwarzl N, Lax S, Uranitsch S, Becker M, Welte Y, Regan JL, Silvestrov M, Kehler I, Fusi A, Kessler T, Herwig R, Landegren U, Wienke D, Nilsson M, Velasco JA, Garin-Chesa P, Reinhard C, Beck S, Schäfer R, Regenbrecht CRA, Henderson D, Lange B, Haybaeck J, Keilholz U, Hoffmann J, Lehrach H, Yaspo LM: Molecular dissection of colorectal cancer in pre-clinical models identifies biomarkers predicting sensitivity to EGFR inhibitors. Nat Commun 8, 14262 (2017).
Die erste Studie, die eine Darmkrebs-Biobank auf Basis von Patientenproben und präklinischen Modellen anlegte sowie ein Datenarchiv zur Medikamentensensitivität bereitstellt, das für eine verbesserte Medikamentenentwicklung und ein besseres Verständnis der Darmkrebs-Biologie genutzt werden kann.
Schumacher D, Andrieux G, Boehnke K, Keil M, Silvestri A, Silvestrov M, Keilholz U, Haybaeck J, Erdmann G, Sachse C, Templin M, Hoffmann J, Boerries M, Schäfer R, Regenbrecht CRA: Heterogeneous pathway activation and drug response modelled in colorectal-tumor-derived 3D cultures. Plos Genet 15, e1008076 (2019).
Diese Arbeit beschreibt, wie Einschränkungen in der auf Genomanalysen basierenden Wirkungsvorhersage von zielgerichteten Therapien mit Hilfe personalisierter Organoid-Modelle überwunden werden können.
Pfohl U, Pflaume A, Regenbrecht M, Finkler S, Graf Ademann Q, Reinhard C, Regenbrecht CRA, Wedeken L: Precision Oncology Beyond Genomics: The Future Is Here – It is Just Not Evenly Distributed. Cells 10, 4 (2021).
Übersichtsarbeit, die aufzeigt wie Multi-omics-Technologien und geeignete In-vitro-Modelle genutzt werden können, um die Tumorheterogenität zu bestimmen und darauf basierend die klinische Praxis im Sinne der modernen Präzisionsonkologie zu verbessern.
Dahlmann M, Gambara G, Brzezicha B, Popp O, Pachmayr E, Wedeken L, Pflaume A, Mokritzkij M, Gül-Klein S, Brandl A, Schweiger-Eisbacher C, Mertins P, Hoffmann J, Keilholz U, Walther W, Regenbrecht C, Rau B, Stein U: Peritoneal metastasis of colorectal cancer (pmCRC): identification of predictive molecular signatures by a novel preclinical platform of matching pmCRC PDX/PD3D models. Molecular Cancer 20, 129 (2021).
Die Studie berichtet zum ersten Mal über die Etablierung personalisierter PDX/PD3D®-Modelle von peritonealen Metastasen kolorektaler Karzinome und stellt eine gründliche molekulare Charakterisierung der Modelle zur Verfügung.
Sankarasubramanian S, Pfohl U, Regenbrecht CRA, Reinhard C, Wedeken L: Context Matters - Why We Need to Change From a One Size Fits all Approach to Made-to-Measure Therapies for Individual Patients With Pancreatic Cancer. Front Cell Dev Biol 9, 760705 (2021).
Die Übersichtsarbeit diskutiert das Reverse Clinical Engineering® als vielversprechende Methode in der personalisierten Medizin zur Bekämpfung tödlicher Pankreaskarzinome.
Wensink GE, Elias SG, Mullenders J, Koopman M, Boj SF, Kranenburg OW, Roodhart JML: Patient-derived organoids as a predictive biomarker for treatment response in cancer patients. Npj Precis Oncol 5, 30 (2011).
Diese Metaanalyse untersuchte die Validität und den klinischen Nutzen von Patienten-abgeleiteten Tumororganoiden zur Bestimmung des individuellen Tumoransprechens.
Brock A, Huang S: Precision Oncology: Between Vaguely Right and Precisely Wrong. Cancer Res 77, 23 (2017).
Die Übersichtsarbeit fasst nicht-genetische Mechanismen der Heterogenität von Tumorzellpopulationen, der Plastizität von Tumorzellen und der Kommunikation zwischen Tumorzellen zusammen, die über den klinischen Erfolg der Präzisionsonkologie entscheiden können.
Kapalczy´nska M, Kolenda T, Przybyla W, Zajaczkowska M, Teresiak A, Filas V, Ibis M, Bli´zniak R, Luczewski L, Lamparska K: 2D and 3D cell cultures - a comparison of different types of cancer cell cultures. Arch Med Sci 14, 4 (2018).
Gegenüberstellung von 2D und 3D-Zellkulturmodellen, welche potenziell besser für die Suche nach neuen Biomarkern und neuen Behandlungsstrategien im Sinne der personalisierten Medizin geeignet sind.
Malone ER, Oliva M, Sabatini PJB, Stockley TL, Siu LL: Molecular profiling for precision cancer therapies. Genome Med 12, 1 (2020).
Die Übersichtsarbeit fasst aktuelle und zukünftige Ansätze zur Umsetzung der Präzisionskrebsmedizin zusammen und zeigt Herausforderungen und potenzielle Lösungen auf, um die Interpretation der Ergebnisse der molekularen Genomanalyse zu erleichtern und ihren klinischen Nutzen zu maximieren.
ASC Oncology
Newsletter
Funktional Immer aktiv
Vorlieben
Statistiken
Marketing
Abonnieren Sie unseren Newsletter
Sie erhalten aktuelle Artikel und Neuigkeiten.

